一种过滤核苷酸多序列比对矩阵中低质量序列的方法
本发明属于系统发育基因组学、生物信息学和比较基因组学,用于dna或rna测序筛选获得的同源序列比对矩阵质量过滤。
背景技术:
1、多序列比对(multiple sequences alignment,msa)是开展进化生物学和生物信息学等研究的基本步骤,可用于构建系统树、选择压力(kaks)分析、基因家族分析等比较基因组和进化研究。同源序列的正确比对,即位置同源性的确定,一直是一项具有挑战性的任务,因为现有的比对方法使用的启发式算法通常无法获得最优解,或存在多个最优解,而且,真实的比对可能在所使用的算法下是次优的。此外,许多因素,如深度差异、插入、倒位、非同源重组、序列的错误组装或注释,以及大数据量,都可能加大正确比对的难度并引起比对错误,尽管其中一些比对软件在某些情况下可能优于其他比对软件。因此,比对问题或多或少不可避免,特别是在基因组学时代,成千上万的比对矩阵无法一一进行人工检查校正。同源多序列比对矩阵中常存在比对错误或存疑的区段、缺失(gap)区段、序列长度过短和整个矩阵长度很短以及有时引入了反向互补序列的情况。现有msa质量过滤的软件如gblock、trimal、bmge、hmmcleaner、taper、spruceup和guidance2通常基于序列相似性或多重比对稳定性来判定msa比对正确性。其中,gblocks、trimal和hmmcleaner就根据序列变异(有时包含缺失gap的频率),将msa中过度变异列、行或整条序列识别为不可靠的部分,这些部分的变异度在统计上落入随机比对或离群范围。变异度过高的列、行或整条序列被过滤掉,而保留其余变异度较低的部分,以便进行下游分析。另一种是通过一组序列在不同比对设置下的比对稳定性/一致性,例如通过设置不同的起始树、比对顺序或子集、序列的头尾(hot)方向或缺失/替换罚分参数,进行多次比对,统计给定的参考矩阵(reference msa;或基础矩阵base msa)在多次比对产生的矩阵中各列、各残基对、或整个矩阵的稳定值(如残基对总分spc、列总分tc),并以此在后续分析中给予不同列不同权重值,或直接过滤掉低稳定列或片段用于后续分析。
2、上述软件虽然能一定程度处理比对错误或存疑的区段、缺失(gap)区段的情况,但目前都存在处理效果不佳、不识别反向互补序列,不识别短序列和短矩阵、对矩阵首尾两端和中间的gaps区段不能分开处理、通常也不能灵活给定阈值。基于列过虑(block-filtering)的软件,如gblock、trimal、bmge等会删除该列中比对正确的序列位点和对占比很低序列比对错误(极少数序列)通常不能识别;基于片段过滤(segment-filtering)的软件,如hmmcleaner的4个意义不明确参数共同控制严格度,难以预测和设置,taper软件难以识别短的、长的和高频率的错误比对,spruceup软件灵敏度和特异性受比对错误程度背景影响;基于列、片段或序列过滤或权重(segment-,block-,or sequence filtering orweighting)的软件,如guidance2如何设置适合的比对条件(不同起始树、替换与插入缺失参数等变化范围设置)、灵敏度和特异性等需检验和探索、且运算量通常很大(时间很久)。而且,仍缺乏对纠正反向互补序列、删除短序列和短矩阵的程序软件。因此,需要开发更全面的程序来解决不同的比对问题,尤其过滤高度变异片段。
技术实现思路
1、为解决上述技术问题,本发明提出了一种过滤核苷酸多序列比对矩阵中低质量序列的方法,显著提升了数据过滤质量,获得更可靠的比对矩阵,为大量相关研究提出了警示和参考,并为保障核苷酸数据分析基本步骤的正确性提供了新方法。
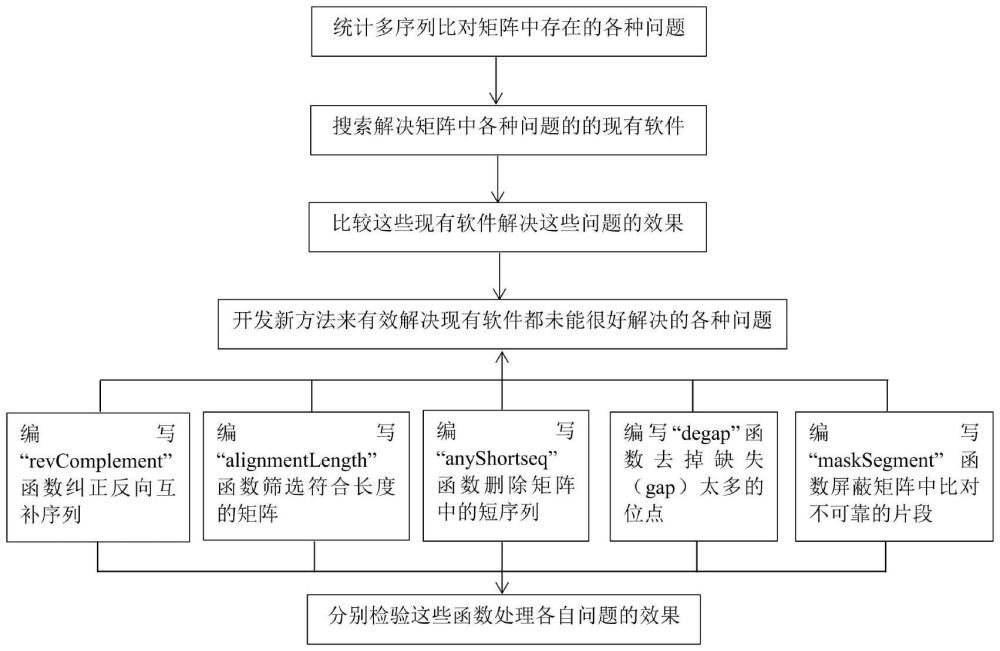
2、为实现上述目的,本发明提供了一种过滤核苷酸多序列比对矩阵中低质量序列的方法,包括:获取核苷酸多序列比对矩阵,对核苷酸多序列比对矩阵筛选出需要反向互补序列,符合长度阈值的矩阵,矩阵中存在的短序列,矩阵中有缺失的列,高度变异比对不可靠的序列这五种低质量序列;对这五种低质量序列进行过滤。
3、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,基于r语言编写的5个函数分别为revcomplement函数、alignmentlength函数、anyshortseq函数、degap函数、masksegment函数;
4、所述revcomplement函数,用于纠正矩阵中反向互补序列;
5、所述alignmentlength函数,用于筛选符合长度阈值的矩阵;
6、所述anyshortseq函数,用于删除矩阵中存在的短序列;
7、所述degap函数,用于删除矩阵中缺失达到预设阈值的列;
8、所述masksegment函数,用于屏蔽矩阵中高度变异、比对不可靠的片段。
9、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,利用所述revcomplement函数纠正矩阵中反向互补序列的方法包括:通过将每个矩阵中的一条序列与其余所有序列进行相似性搜索,获得相似性搜索结果文件;
10、根据相似性搜索结果文件中的strand=plus/minus判断矩判断为方向不一致,找到存在反向互补序列的矩阵;
11、通过判断矩阵中strand=plus/minus的序列数目和strand=plus/plus的序列数目,将数目少的这组序列,在矩阵中将进行反向互补,重新输出矩阵并放在一个新文件夹中,不存在反向互补序列的矩阵选择输出在另一个新文件夹中。
12、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,利用所述alignmentlength函数筛选符合长度阈值的矩阵的方法包括:通过直接判断矩阵总长度,矩阵总长度大于等于设置阈值长度时,将矩阵输出在一个新文件夹中;矩阵总长度小于设置阈值长度的矩阵选择输出在一个新文件夹中。
13、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,利用所述anyshortseq函数删除矩阵中存在的短序列的方法包括:
14、提取矩阵中的每条序列名称和对应的碱基序列,判断序列的碱基长度并与设置的阈值进行比较;
15、当序列碱基长度小于设置阈值长度时将矩阵中的序列删掉并将矩阵输出在一个新文件夹中,碱基长度大于等于设置阈值的矩阵选择输出在一个新文件夹中;
16、用户根据自身数据特点和需求设置阈值,所述阈值按碱基长度或碱基长度占序列全长比例的两种方式设置,这两种设置方式是或者的关系,属于情况之一就删掉。
17、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,利用所述degap函数删除矩阵中缺失区段的方法包括:
18、对矩阵两端和中间位置缺失位点分别设置参数阈值处理,
19、对两端缺失位点进行处理,根据阈值判断前端小于阈值的位点并删除,判断末端小于阈值的位点并删除;
20、对整个矩阵的缺失位点进行处理,根据设置的阈值判断小于阈值的位点并删除;
21、删除缺失位点的矩阵输出在一个新的文件夹,未删除缺失位点的矩阵选择输出在另一个新的文件夹。
22、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,利用所述masksegment函数屏蔽矩阵中高度变异、比对不可靠的片段的方法包括:
23、计算随机相似性概率与随机排列相似性阈值之间的相关性;
24、提取比对好矩阵中每条序列名称和碱基序列形成完整的比对序列;
25、将所述比对序列按设定的窗口大小和步长划分成窗口;
26、计算成对相似性得分,并与随机排列相似性阈值比较,进行分组和合并,将符合条件的片段归入特定的组中;
27、将每个组内的片段,根据相似性得分与预设的随机排列相似性阈值的比较来确定是否需要标记为需要屏蔽的片段。
28、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,计算随机相似性概率p的方法为:
29、
30、其中,n表示片段长度;k表示替换或变异位点的数量。
31、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,计算随机排列相似性rs的方法为:
32、
33、其中,n表示片段长度;k表示替换或变异位点的数量。
34、本发明提供的过滤核苷酸多序列比对矩阵中低质量序列的方法,计算成对相似性得分ps的方法为:
35、
36、其中,n表示片段长度;k表示替换或变异位点的数量;xi和yi表示两条不同序列的滑动窗口中的每对片段。
37、本发明技术效果:本发明公开了一种过滤核苷酸多序列比对矩阵中低质量序列的方法,基于研究中发现的多序列比对矩阵中存在的各种比对问题,通过比较现有过滤方法的优缺点,并用实际数据检验现有软件的过滤效果后发现,处理效果不佳或能设置的阈值有限,而且,仍缺乏对纠正反向互补序列、删除短序列和短矩阵的程序软件。因此本发明开发了一套处理函数分别处理各种比对问题,其中的核心函数masksegment是通过滑窗方式,根据序列变异程度,采用分组-重组(gruoping-regruoping)的方法来过滤矩阵中错误比对或存疑片段,与现有软件方法不同。通过同样的数据,不同的比对错误形式(高频和低频错误比对、长片段和段片段错误比对),比较了本发明与现有方法对错误比对或存疑片段的处理效果,结果表明:在高频和低频错误比对、长片段和段片段错误比对等情形下,masksegment函数的过滤效果都更好。另外,现有矩阵优化软件未包括处理矩阵(或未比对序列数据)中反向互补序列、识别短矩阵的功能,以及对空位(gap)的处理方式不够全面、对删除短序列的方法不够简单直接等问题。本发明都分别开发有函数专门且有效的处理这些问题,而且操作简单可控性强,具体阈值可以由用户根据需求自由设定。
技术研发人员:张强,覃信梅,卢永彬,李鹏伟,羊学荣,黄夕洋
技术所有人:广西壮族自治区中国科学院广西植物研究所
备 注:该技术已申请专利,仅供学习研究,如用于商业用途,请联系技术所有人。
声 明 :此信息收集于网络,如果你是此专利的发明人不想本网站收录此信息请联系我们,我们会在第一时间删除
