一种基于深度学习和长读测序的SNP和INDEL检测方法
本发明涉及生物信息学,具体是一种基于深度学习和长读测序的snp和indel检测方法。
背景技术:
1、单核苷酸多态性(snp)和插入/缺失(indel)是人类基因组中两种常见的遗传变异,与人类遗传疾病息息相关;而利用高效的snp和indel检测算法,可以在个体基因组中识别与特定遗传疾病相关的变异,有助于疾病进行诊断和筛查。
2、目前业内普遍依赖传统的短读测序方法,在灵活性与泛化能力、计算效率上仍然落后,并且处理复杂问题时所需要的计算成本过于高昂,而且短读测序在面对复杂基因组时仍存在许多难以检测的区域。
技术实现思路
1、本发明实施例的目的在于提供一种基于深度学习和长读测序的snp和indel检测方法,该方法使用深度学习技术和长读测序技术,解决传统相关技术在灵活性、泛化能力、计算效率上的低性能,以及面对复杂问题时短读测序存在的无法检测区域的技术问题。
2、为实现上述目的,本发明提供了如下的技术方案。
3、本发明一实施例提供了一种基于深度学习和长读测序的snp和indel检测方法,包括:
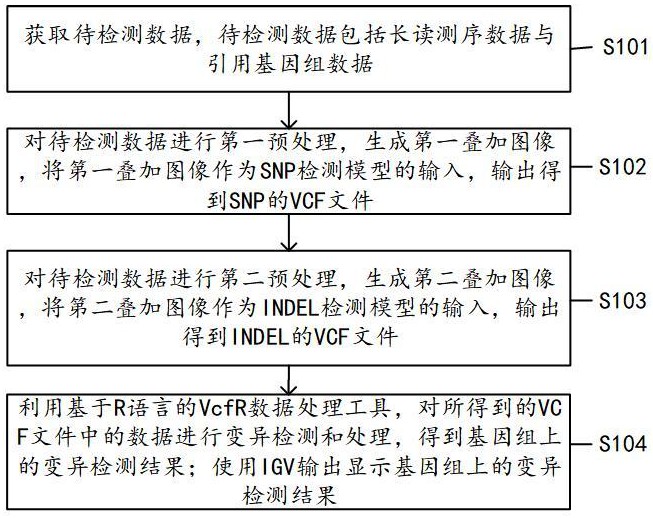
4、获取待检测数据,待检测数据包括长读测序数据与引用基因组数据;
5、对待检测数据进行第一预处理,生成第一叠加图像,将第一叠加图像作为snp检测模型的输入,输出得到snp的vcf文件;
6、对待检测数据进行第二预处理,生成第二叠加图像,将第二叠加图像作为indel检测模型的输入,输出得到indel的vcf文件;
7、利用基于r语言的vcfr数据处理工具,对所得到的vcf文件中的数据进行变异检测和处理,得到基因组上的变异检测结果;
8、使用igv输出显示基因组上的变异检测结果。
9、进一步的,所述对待检测数据进行第一预处理,生成第一叠加图像的步骤,包括选择候选位点的过程;其中:
10、设定一个参考碱基为r的基因组位点b,计算覆盖度,覆盖度的计算公式表示为:
11、;
12、式中,n表示覆盖基因组位点b的读段数量;表示第i个读段的长读,l表示目标区域的长度;
13、在当前候选点位的覆盖度低于覆盖度阈值(阈值设为10)时,将当前候选点位去除,得到snp候选位点的子集,并将该子集作为杂合snp位点集v;
14、还包括生成单倍型snp的叠加图像的过程,其中:
15、对于snp候选位点b,从位点集v中选择与候选位点b共享至少一个读取且距离候选位点b最多50000bp的站点;
16、在候选位点b的每个方向,从位点集v中选择20个站点,用z来表示候选位点b及候选位点b附近潜在杂合snp位点的集合;
17、将覆盖候选位点b的读集分为四组,其中,={在b处支持碱基b的读},b∈{a,g,t,c},不使用不支持候选位点b上任何碱基的读;
18、对于中每个支持碱基为b的读组,计算碱基为d∈{a,g,t,c}的位点t∈z的支持读组个数;设,其中,表示一变量,是一个函数,如果d是t点的参考基,则返回−1,否则返回1;进而得到一个4×41×4三维矩阵m,其中三维矩阵m的条目表示为,三维矩阵m的第一维对应b位点的核苷酸类型b,第二维对应于t位点的数量,第三维对应于t位点的核苷酸类型d;
19、三维矩阵m的图像以读段组为行,各种碱基位置为列,有4个通道,每个通道记录了给定位点上给定读段组中不同碱基的频率;
20、将另一个通道添加到图像中,新加入的图像为一个4×41矩阵,元素数量为,如果碱基b是位点b的参考碱基,则,否则为0;在这个通道中,有一行1作为b点的参考基,一行0作为其他基;先对集合z中的基点参考碱基进行编码,再将编码后得到的结果作为新的一行加入图像中,得到叠加图像。
21、进一步的,所述snp检测模型基于卷积神经网络cnn构建,包括三层卷积层:
22、第一层卷积层使用三个不同维度的核,并将卷积特征组合成单个输出;所述三个不同维度的核中的一个从一行捕获局部信息,另一个从一列捕获局部信息,最后一个从二维局部区域捕获局部信息;
23、第二层卷积层和第三层卷积层均使用大小为2×3的核;第三层卷积层的输出被扁平化,并用作具有dropout的全连接网络的输入;
24、所述全连接网络第一个全连接层之后是两个不同的全连接层的神经网络,用于以计算两种类型的概率;其中,在第一个神经网络中,计算每个b型核苷酸的概率,以表明b存在于基因组候选位点,对于每个b型核苷酸,都有一个二值标签预测;第二个神经网络将第一个神经网络的logit输出与第一个全连接隐藏层的输出相结合,用于估计候选位点合子性的概率。
25、进一步的,所述估计候选位点合子性的概率的步骤,包括:
26、利用神经网络计算每种核苷酸类型在候选位点存在的概率;
27、如果一个候选位点至少有两种概率超过0.5的核苷酸类型,则认为它是杂合的;否则认为是纯合的;
28、对于杂合位点,用杂合变体选择两种概率最高的核苷酸类型;对于纯合子位点,只选择概率最高的核苷酸类型:如果该核苷酸类型不是参考等位基因,则进行纯合子变体调用;否则,则为纯合-参照;
29、其中,每个变体调用被分配一个质量分数,计算公式表示为:
30、;
31、式中,表示替代等位基因b的概率,并记录为vcf文件的qual字段中的浮点数,用于指示假阳性预测的机会;分数越大,预测错误的可能性越小。
32、进一步的,所述输出得到indel的vcf文件的步骤,包括:
33、使用whatshap预测snp调用;
34、输出由snp检测模型生成的无相位vcf文件和由whatshap生成的相位vcf文件。
35、进一步的,所述对待检测数据进行第二预处理,生成第二叠加图像的步骤,包括选择候选点位和生成叠加图像,其中,在选择候选点位之前,包括对待检测数据进行质量过滤的过程,该过程如下:
36、使用phred质量评分过滤低质量碱基,phred质量评分计算公式表示为:
37、;
38、式中,p是碱基调用的错误概率,当质量评分低于阈值时,舍去该碱基;所述阈值设置为20;
39、去除测序过程中引入的适配子序列,以避免干扰后续分析;
40、去除pcr扩增过程中产生的重复读取,以减少偏倚;
41、将读取与参考基因组对齐,并去除未对齐或对齐质量低的读取;
42、去除过短或过长的读取,以确保读取长度的一致性和分析的准确性;
43、去除包含大量重复序列或低复杂度区域的读取。
44、进一步的,所述第二预处理中选择候选点位的步骤,包括:
45、对于i∈{0,1},计算以下三个数据:
46、数据一:=位点b的第i阶段读数总数;
47、数据二:插入频率=;
48、数据三:删除频率=;
49、当出现以下两种情况时,b被认为是indel候选地点:
50、和均大于指定的深度阈值;
51、插入频率大于指定的插入频率阈值,或删除频率大于指定的删除频率阈值。
52、进一步的,所述第二预处理中生成叠加图像的步骤,包括:
53、对于一个indel候选站点b,用、和分别表示所有读取的集合,读取在一个相位,读取在另一个相位;
54、设为从site b开始,长度为160bp的参考序列;
55、对于每个集合,执行如下操作:对于每个读段r∈s,设为从站点b开始的读段的160bp长的子序列;使用muscle对以下一组序列进行多重序列比对;设为重新比对的序列,其中表示重新比对的参考序列,表示序列的重新比对;从末端开始截断所有长度为128的序列;对于b∈{a,g,t,c,−}和1≤p≤128,计算:
56、;
57、;
58、式中,表示一个函数,其中,如果的索引p处的基数是b,则返回1,否则返回0;矩阵c表示原始计数,p表示每个符号;m为包含条目的5×128矩阵;
59、构造一个5×128矩阵q,其条目为,如果在索引p处有符号b,则,否则为0;矩阵m和q都有第一维对应符号{a,g,t,c,−},第二个维度对应重新排列序列的堆积列;
60、构造一个5×128 ×2矩阵,其第一通道为矩阵m−q,图像实际上就是一个二维矩阵,这里得到的为一个三维矩阵,可以看成是一个叠第二通道为矩阵q;
61、拼接三个矩阵、和一起得到一个15×128×2矩阵作为检测模型的输入叠加图像。
62、进一步的,所述的indel检测模型基于卷积神经网络cnn构建,包括三层卷积层:
63、第一层卷积层使用三个不同维度的核,并将卷积特征组合成单个输出;所述三个不同维度的核中的一个从一行捕获局部信息,另一个从一列捕获局部信息,最后一个从二维局部区域捕获局部信息;
64、第二层卷积层和第三层卷积层均使用大小为2×3的核;第三层卷积层的输出被扁平化,并用作具有dropout的全连接网络的输入;
65、所述全连接网络的第一个完全连接层之后是两个完全连接的隐藏层,用于产生候选位点上四种合子情况的概率估计:纯合子-参考、纯合子-替代、杂合子-参考和杂合子-替代。
66、进一步的,所述将第二叠加图像作为indel检测模型的输入,输出得到indel的vcf文件的步骤,包括:
67、计算四种合子的概率;如果纯合-参考标记具有最高的概率,则不进行变体调用;如果纯合-备选标签具有最高的概率,从的多序列比对中确定共识序列,并使用bio-python的pairwis2具有仿射间隔惩罚的全局比对算法在候选位点与参考序列进行比对;
68、替代等位基因是由两个序列的成对比对中的alignmnet gaps推断出来的;
69、在杂合预测的任何一个具有最高概率的情况下,使用和分别确定每个相的一致性序列,并将其与参考序列进行比对,两个阶段的indel检测结果被结合起来,在候选位点导出最终的分阶段indel检测结果,根据检测结果创建indel的vcf文件。
70、与现有技术相比,本发明基于深度学习和长读测序的snp和indel检测方法的有益效果是:
71、第一,本发明使用了更加适配的预处理方法,具体的,使用覆盖度计算和phred质量评分两种数据预处理方法进行数据的预处理,能够更好的过滤掉低质量的位点并筛选出具有更高检测潜力的位点,可以明显提高snp和indel检测的准确性;
72、第二,本发明与其他长读变体调用者相比,可以在复杂的基因组区域产生snp/indel检测,利用重复区域长读数据的优势,能够检测短读测序技术无法可靠检测到的高置信度区域外的snp/indels,从而为短读测序未发现病因候选变异的未诊断疾病的因果变异研究提供更多的候选snp/indels位点;
73、第三,本发明通过构建候选位点的堆叠图像,并使用卷积神经网络进行特征提取和分类,以提高snp和indel检测的准确性和效率;本发明通过利用长读测序数据覆盖大范围基因组区域,提高基因组装的连续性和准确性,解决使用寻常短读测序方法面临复杂问题时遇到的难题。
74、综上所述,本发明使用了深度学习技术和长读测序技术,能够很好的解决传统方法在灵活性与泛化能力、计算效率上的低性能和面对复杂问题时短读测序存在的无法检测区域的问题。
技术研发人员:石博艺,王永梅,王芃力,吴雨涵,潘海瑞,陈文希,张奥运,范子建
技术所有人:安徽农业大学
备 注:该技术已申请专利,仅供学习研究,如用于商业用途,请联系技术所有人。
声 明 :此信息收集于网络,如果你是此专利的发明人不想本网站收录此信息请联系我们,我们会在第一时间删除
