基于物理学捕获循环肿瘤细胞的全基因组数据分析方法与流程
本发明属于生物信息学,具体涉及一种基于物理学捕获循环肿瘤细胞的全基因组数据分析方法。
背景技术:
1、循环肿瘤细胞(circulating tumor cells, ctcs)是从原发肿瘤或转移肿瘤脱落进入血液循环的肿瘤细胞,这些肿瘤细胞进入血液后大部分受机体免疫机制或流体剪切力等因素被清除,少数逃逸存留的细胞可在远处种植并发展为转移肿瘤,明显加剧恶性肿瘤患者死亡风险。目前,循环肿瘤细胞作为主要的液体活检标志物之一,常规ctc计数在肿瘤的早期诊断、疗效评估、预后预测、复发转移监测等方面发挥重要作用。随着肿瘤研究的深入,ctc的应用方向不断拓展,其下游分析也颇受关注,如ctc分子分型、基因检测、细胞培养等,这些研究可提供临床除计数之外更为丰富的肿瘤信息,对于指导患者用药、肿瘤耐药及转移机制研究具有较高的价值。
2、目前市面上存在的循环肿瘤细胞分离和鉴定方法有许多种。分离方法主要包括基于免疫学原理和物理学原理两大类。免疫学方法是指根据细胞表面标记物(如ck、epcam、vimentin、cd45等)捕获肿瘤细胞,包括阳性富集和阴性富集,此类方法特异性较高,但由于ctc的稀有性和异质性高,且在血液循环中会发生上皮-间质转化(emt),因而阳性富集可能会遗漏部分emt转化的肿瘤细胞;阴性富集通过滤除白细胞以达到肿瘤细胞富集目的,操作步骤较多,需考虑细胞损失和富集纯度的问题。物理学方法根据细胞的物理特性(细胞大小过滤、梯度密度离心、细胞电荷差异等)捕获肿瘤细胞,不依赖于细胞标志物,适用大部分实体肿瘤。鉴定方法主要包括细胞形态学鉴定、免疫荧光鉴定、fish鉴定、适配体pcr等。然而,由于肿瘤具有高度异质性,以上常规检测方法均可能存在假阳性或假阴性问题,这在一定程度限制了ctcs的临床应用。
3、二代测序(next generaion sequencing, ngs)是一种高通量测序方法,可以检测包括点突变、插入缺失、拷贝数变异、微卫星不稳定等各种基因变异信息。其中,拷贝数变异(copy number variation, cnv)是由基因组发生重排而导致的,属于一种结构变异形式,分析整体染色体变异情况,且相较于深度基因panel检测价格更低。研究发现,cnv在恶性肿瘤中几乎普遍存在,同类型肿瘤间具有相似的cnv模式,而不同类肿瘤则具有各自的cnv模式,因此cnv检测可作为恶性肿瘤的判断标志。
技术实现思路
1、有鉴于此,本发明旨在开发一种获取循环肿瘤细胞并对其进行测序和分析的方法,以克服循环肿瘤细胞计数检测的应用局限和恶性肿瘤患者无法实时获取组织进行良恶性辨别的问题,以及现有ctc检测方法可能出现假阳性和假阴性的问题,进一步提高肿瘤检测特异性,为临床提供更为精确的肿瘤信息。
2、本发明的技术方案具体如下:
3、本发明提供了一种基于物理学捕获循环肿瘤细胞的全基因组数据分析方法,其包括以下步骤:
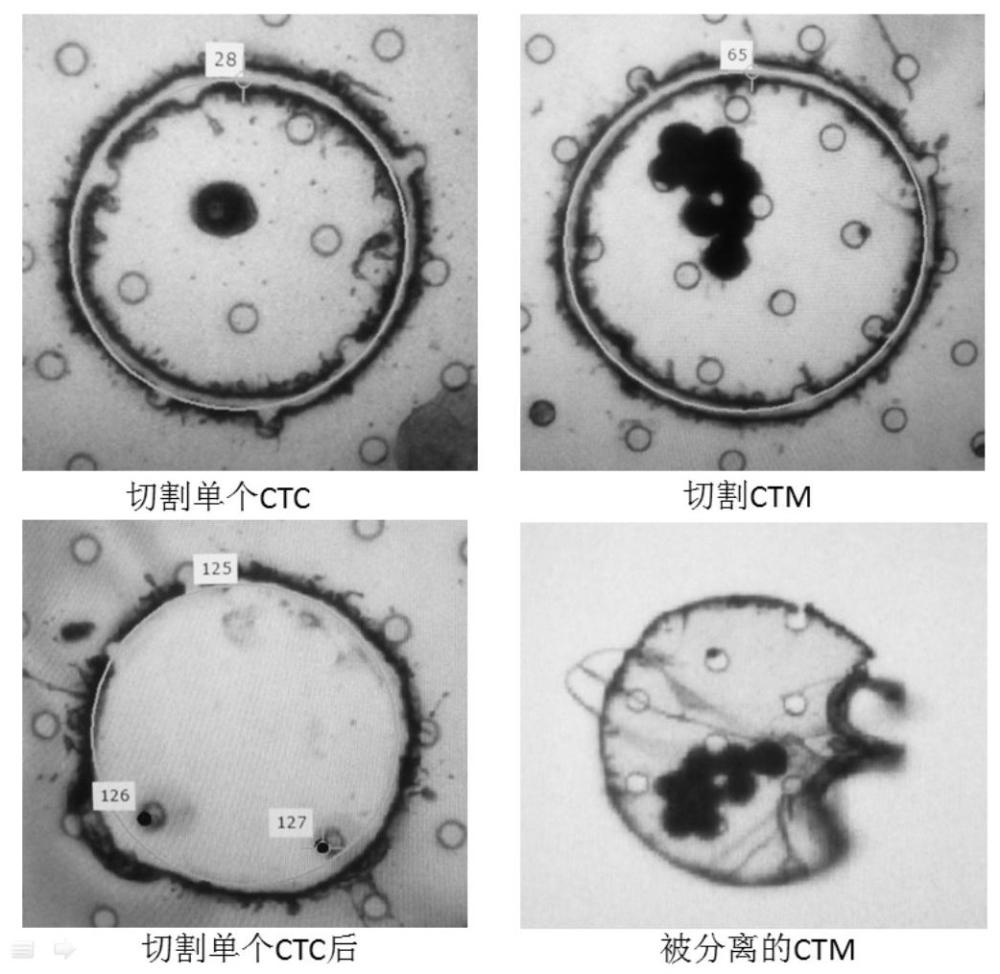
4、s1、采用ctcbiopsy系统基于细胞物理特性分离富集血液样本中的循环肿瘤细胞,根据细胞形态并通过激光显微切割挑取靶细胞;
5、s2、对靶细胞进行全基因组测序;
6、s3、对测序数据分析,以获得靶细胞遗传变异信息。
7、在本发明方法中,循环肿瘤细胞包括但不限于为肺癌、胃癌、结肠腺癌、胶质瘤和乳腺癌等患者血液样本中的循环肿瘤细胞。
8、优选地,在上述方法中,ctcbiopsy系统所用滤膜的孔径为8μm。
9、优选地,在上述方法中,步骤s1包括:
10、s11、取受试患者外周血,利用ctcbiopsy系统进行循环肿瘤细胞的分离富集;
11、s12、将ctcbiopsy系统中的滤膜取出,采用瑞氏-吉姆萨染色液进行细胞染色和制片;
12、s13、阅片并根据细胞形态通过激光显微切割挑取待测靶细胞,其中所述靶细胞的挑取标准至少符合以下任一情形:
13、①核型异常,如胞核呈分叶状、桑葚状等,可对比周围血细胞的形态进行判断;
14、②核浆比大于0.8;
15、③细胞直径(长端)大于15μm;
16、④胞核深染且着色不均匀,主要原因是肿瘤细胞染色质增多,颗粒变粗;
17、⑤核膜增厚,出现凹陷或褶皱,且核膜呈不规则状;
18、⑥出现巨大核仁或异常核分裂。
19、更为优选地,若所述靶细胞符合至少4种情形可确认为ctc,若所述靶细胞具有情形⑥且同时符合其他两种情形可确认为ctc,若所述靶细胞仅具有情形⑥或符合其他任意三种情形可确认为疑似ctc。其中,由3个或3个以上的ctc聚集在一起所组成的细胞团则确认为循环肿瘤微栓(ctm)。
20、更为优选地,步骤s11具体为:用多聚甲醛和生理盐水对外周血样本进行固定和稀释;使用75%酒精和生理盐水润洗ctcbiopsy系统中的细胞滤器,然后对外周血样本进行分离富集。
21、更为优选地,步骤s12具体为:将细胞滤器中的滤膜取出并贴于玻片上,先用曙红染色,再用亚甲蓝染色。
22、优选地,在上述方法中,步骤s2具体为:先采用malbac全基因组扩增试剂盒进行靶细胞基因组扩增,再利用二代测序技术进行低深度全基因组测序。
23、在上述方法中,步骤s3获得靶细胞遗传变异信息包括但不限于单核苷酸变异(snvs)、插入缺失突变(indels)和cnv等。优选地,靶细胞遗传变异信息为cnv,且步骤s3具体包括以下过程:
24、s31、对测序数据进行质控;
25、s32、使用bowtie2将质控后所得reads与参考基因组比对,使用samtools计算碱基的coverage值,区分不同染色体根据200k长度划分bin单元,计算bin的平均coverage值,根据公式(1)和(2)计算z-score值,再以z-score值进行cnv图谱分析;
26、其中,公式(1)和公式(2)具体如下所示:
27、,
28、,
29、式中,coverageraw是指测序获得的原始覆盖率,coveragecontrols, raw是指实验对照样本的原始覆盖率,vtumor是指肿瘤样本的归一化测序覆盖率,vcontrol为对照样本在的归一化测序覆盖率。
30、更为优选地,步骤s31所述质控的条件为:
31、过滤掉含有接头序列的reads;
32、当单端测序read中含有的未测出碱基数超过该条read长度比例的5%时,过滤掉该read以及跟该read构成paired-end对的read;
33、当单端测序read中含有的低质量碱基数超过该条read长度比例的20%时,过滤掉该read以及跟该read构成paired-end对的read。
34、更为优选地,步骤s32中的参考基因组为人类基因组参考序列grch38。
35、相对于现有技术,本发明的有益效果为:
36、本发明方法基于细胞大小利用ctcbiopsy系统进行ctc分离富集,并采用细胞形态学进行靶细胞挑取,这种方式不依赖于肿瘤细胞表面标记物,对循环肿瘤细胞微栓分离效果更好,且具有操作简便、富集效率高的优势,还可以减少对ctc的损伤以达到保持ctc细胞完整性的目的,便于后续进行靶细胞全基因组测序。
37、本发明方法适用于大多数实体肿瘤,且与多种ctc下游测序、分析方法兼容,包括但不限于ctc-cnv检测、ctc-panel检测、ctc-wes检测、ctc全基因组检测。如在本发明的一些实施例中,对分离的ctc或疑似ctc进行cnv分析,通过ctc全基因组低深度测序数据分析,可获得更精准的肿瘤信息,尤其对于肿瘤的早期诊断识别具有重要意义。
技术研发人员:吴高,杨运强,吴长发,邓钢,徐智高,徐凡,阚光雄,李元娥,黄佳玉,柯晓明,魏亮,姚远
技术所有人:武汉友芝友医疗科技股份有限公司
备 注:该技术已申请专利,仅供学习研究,如用于商业用途,请联系技术所有人。
声 明 :此信息收集于网络,如果你是此专利的发明人不想本网站收录此信息请联系我们,我们会在第一时间删除
